Your job Demo6_Adenine_RIboswitch_no_ligand_DMS_mode (ID: 00000000_000000-DEMO6) is ready!
The results are kept on the server for 1 week. Thank you for using this web server, and keep on keepin' on!
The results are kept on the server for 1 week. Thank you for using this web server, and keep on keepin' on!
| Name | Method | Filenames | Full string | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | Adenine_Riboswitch_DMS_adenine_1_to_DMS_adenine_3_mean_dms_mode | GGGGGCCUUCGGGCCAACGCUUCAUAUAAUCCUAAUGAUAUGGUUUGGGAGUUUCUACCAAGAGCCUUAAACUCUUGAUUAUGAAGUGUCGAUCCGGUUCGCCGGAUCCAAAUCGGGCUUCGGUCCGGUUC | G | G | G | G | G | C | C | U | U | C | G | G | G | C | C | A | A | C | G | C | U | U | C | A | U | A | U | A | A | U | C | C | U | A | A | U | G | A | U | A | U | G | G | U | U | U | G | G | G | A | G | U | U | U | C | U | A | C | C | A | A | G | A | G | C | C | U | U | A | A | A | C | U | C | U | U | G | A | U | U | A | U | G | A | A | G | U | G | U | C | G | A | U | C | C | G | G | U | U | C | G | C | C | G | G | A | U | C | C | A | A | A | U | C | G | G | G | C | U | U | C | G | G | U | C | C | G | G | U | U | C | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | Adenine_Riboswitch_DMS_adenine_1_to_DMS_adenine_3_mean_dms_mode | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | 0.35 | 0.04 | 0.13 | 0.15 | 1.00 | 1.00 | 0.50 | 0.50 | 0.22 | 0.01 | 0.00 | 0.56 | 0.07 | 0.13 | 0.16 | 0.34 | 1.00 | 1.00 | 0.20 | 0.12 | 0.03 | 0.05 | 0.02 | 0.12 | 0.01 | 0.02 | 0.06 | 0.18 | 0.17 | 0.25 | 0.08 | 0.02 | 0.00 | 0.00 | 0.00 | 0.00 | 0.09 | 0.06 | 0.23 | 0.00 | 0.00 | 0.03 | 0.03 | 0.30 | 0.27 | 0.30 | 0.07 | 0.40 | 0.22 | 0.16 | 0.00 | 0.19 | 0.00 | 0.03 | 0.07 | 0.15 | 0.11 | 0.56 | 0.36 | 0.50 | 0.03 | 0.01 | 0.01 | 0.04 | 0.14 | 0.58 | 1.00 | 0.06 | 0.41 | 0.17 | 0.04 | 0.04 | 0.10 | 0.17 | 0.08 | 0.00 | 0.04 | 0.03 | 0.18 | 0.00 | 0.09 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | -1.00 | ||
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | RNAstructure_Fold_DMS | Adenine_Riboswitch_DMS_adenine_1_to_DMS_adenine_3_mean_dms_mode | ...((((....)))).((((((((((...(((((((......)).)))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | ) | ) | . | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | RNAfold | ...((((....)))).((((((((((...((((((.........))))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | RNAstructure_ProbKnot | ...((((....)))).((((((((((...(((((((......).))))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | ) | . | ) | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | RNAstructure_Fold | ...((((....)))).((((((((((...((((((.........))))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | CentroidFold | ...((((....)))).((((((((((....(((((.........))))).........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | CONTRAfold | ...((((....)))).((((((((((..((((((((......).))))))((....))((((((.......)))))).))))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | ) | . | ) | ) | ) | ) | ) | ) | ( | ( | . | . | . | . | ) | ) | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | IPknot | ...((((....)))).((((((((((....(((((.........))))).........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | Linearfold_C | ...((((....)))).((((((((((...((((((.........))))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | Linearfold_V | ...((((....)))).((((((((((...((((((.........))))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . | |
| Demo6_Adenine_RIboswitch_no_ligand_DMS_mode | Consensus_from_predictions | ...((((....)))).((((((((((...((((((.........))))))........((((((.......))))))..)))))))))).(((((((....)))))))..((((((((....)))))))). | . | . | . | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | . | . | . | . | . | . | ( | ( | ( | ( | ( | ( | . | . | . | . | . | . | . | ) | ) | ) | ) | ) | ) | . | . | ) | ) | ) | ) | ) | ) | ) | ) | ) | ) | . | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | . | . | ( | ( | ( | ( | ( | ( | ( | ( | . | . | . | . | ) | ) | ) | ) | ) | ) | ) | ) | . |
Secondary structure predictions
Fold predicted structure, based on DMS reactivities:
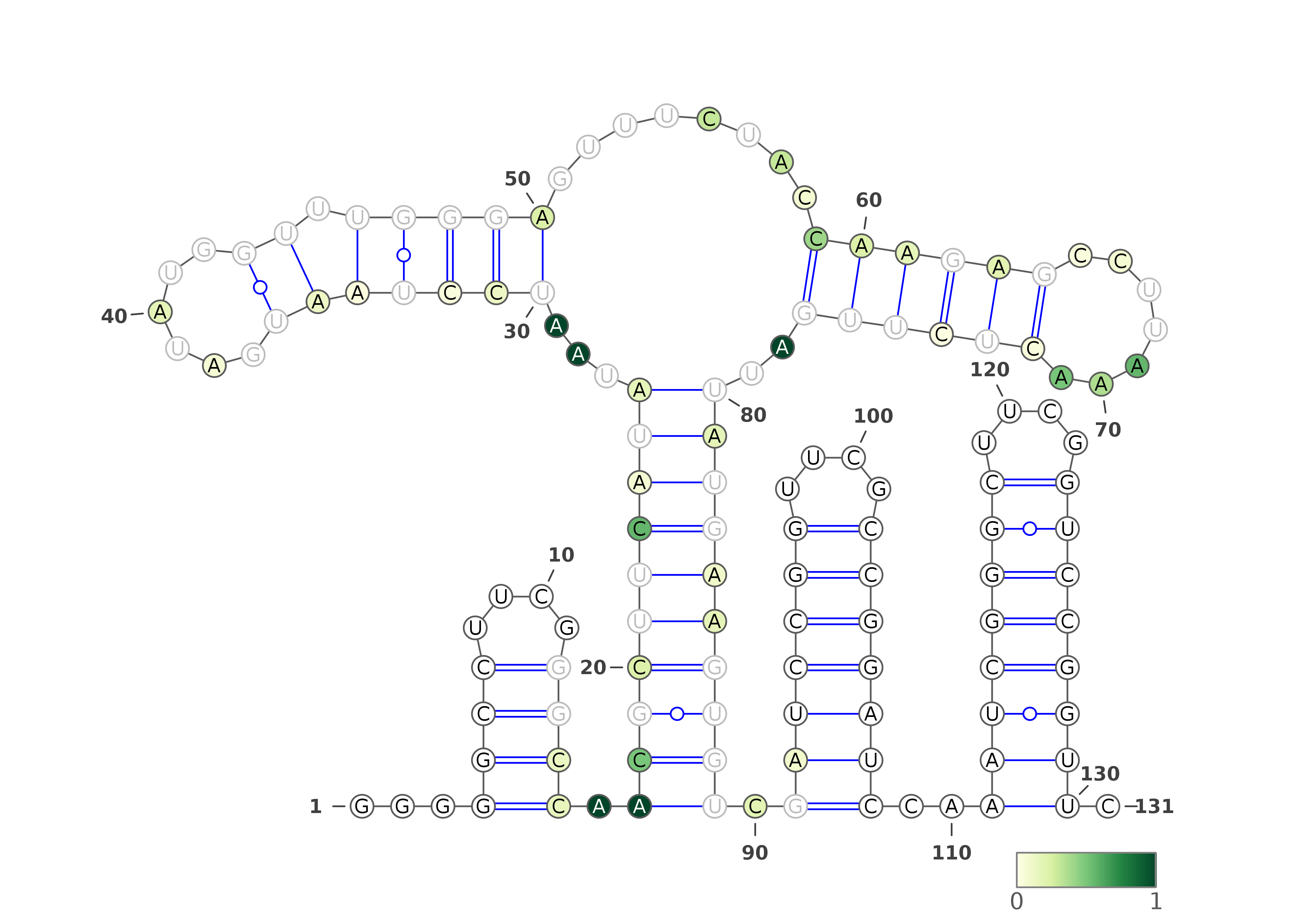
Fold predicted structure, based on DMS reactivities - radiate representation:
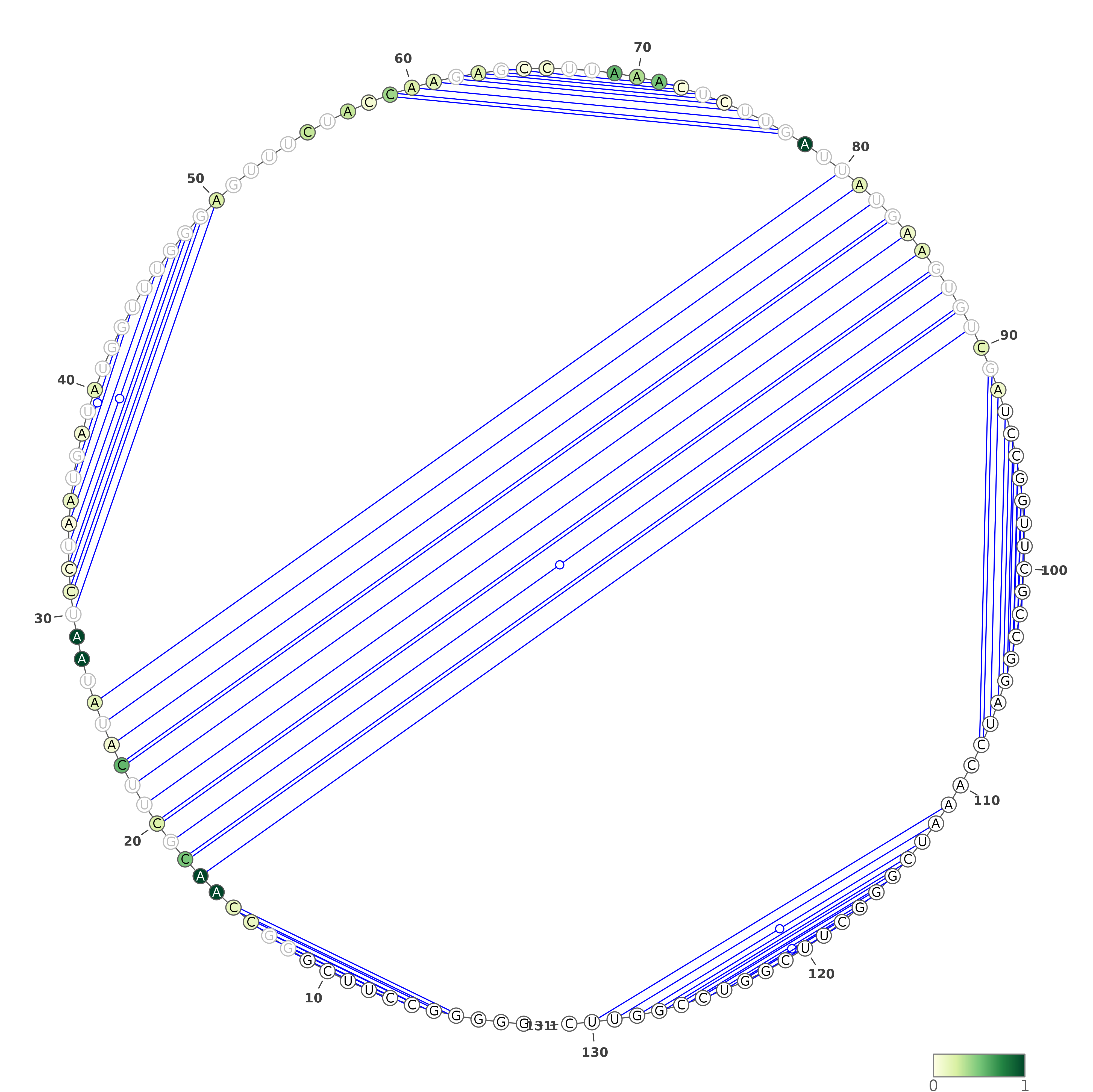
Fold predicted structure, based on DMS reactivities - circular representation:
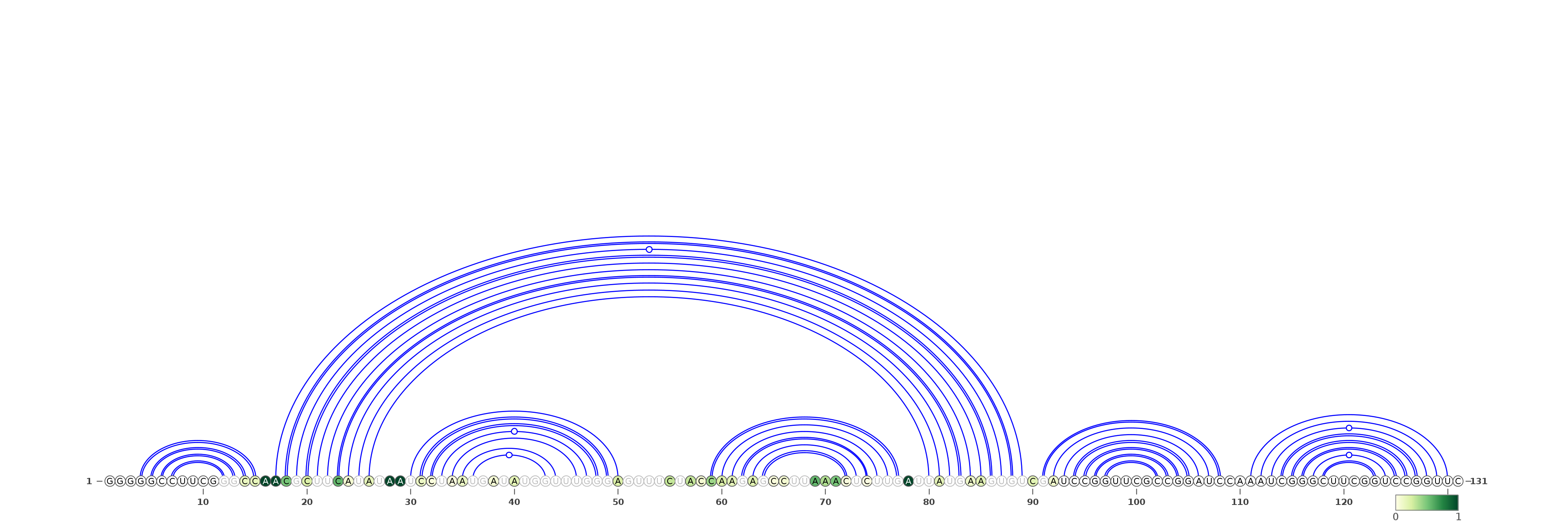
Fold predicted structure, based on DMS reactivities - linear representation
Other secondary structure predictions methods and visualisations
Reactivity mapping to structures predicted by other methods are also ready to display or download. Secondary structure to display and copy to clipboard, links to external tools (SimRNAweb )
| method | radiate | circular | line | secondary structure | copy | SimRNAweb |
|---|---|---|---|---|---|---|
| RNAfold | display | | display | | display | | Predict 3D structure for this SS | ||
| ProbKnot | display | | display | | display | | Predict 3D structure for this SS | ||
| Fold | display | | display | | display | | Predict 3D structure for this SS | ||
| CentroidFold | display | | display | | display | | Predict 3D structure for this SS | ||
| CONTRAfold | display | | display | | display | | Predict 3D structure for this SS | ||
| IPknot | display | | display | | display | | Predict 3D structure for this SS | ||
| LinearFold_C | display | | display | | display | | Predict 3D structure for this SS | ||
| LinearFold_V | display | | display | | display | | Predict 3D structure for this SS | ||
| Consensus | display | | display | | display | | Predict 3D structure for this SS |
If you use this software please cite as:
Tomasz K. Wirecki, Katarzyna Merdas, Agata Bernat, Michał J. Boniecki, Janusz M. Bujnicki, and Filip Stefaniak, RNAProbe - a web server for normalization and analysis of RNA structure probing data. Nucleic Acids Research, Volume 48, Issue W1, 02 July 2020, Pages W292–W299, https://doi.org/10.1093/nar/gkaa396
Tomasz K. Wirecki, Katarzyna Merdas, Agata Bernat, Michał J. Boniecki, Janusz M. Bujnicki, and Filip Stefaniak, RNAProbe - a web server for normalization and analysis of RNA structure probing data. Nucleic Acids Research, Volume 48, Issue W1, 02 July 2020, Pages W292–W299, https://doi.org/10.1093/nar/gkaa396
Analysis mode: dms; outfilename core: Adenine_Riboswitch_DMS_adenine_1_to_DMS_adenine_3_mean_dms_mode.